MAINZ – BMBF fördert Forschungsprojekt zur Entwicklung einer digitalen Benutzeroberfläche für hocheffiziente Gewebeuntersuchung auf der Basis von künstlicher Intelligenz
(Mainz, 23. Januar 2019, ok) Eine spezielle Softwareplattform zu entwickeln, mit der sich schnell und präzise entscheiden lässt, ob Gewebe Tumorzellen enthält oder nicht, das ist das Ziel eines neuen Forschungsprojekts an der Universitätsmedizin Mainz. Das BMBF fördert dieses Projekt mit knapp einer halben Millionen Euro über einen Zeitraum von zwei Jahren.
Gibt es Anhaltspunkte für tumorverdächtige Gewebeareale im Körper eines Patienten? Liegt ein Tumor vor oder nicht? Inwieweit weist das zu analysierende Gewebe relevante genetische Veränderungen auf? Mit welchen Therapieoptionen lässt sich ein identifizierter Tumor potentiell bekämpfen? Antworten auf diese und vergleichbare Fragen soll ein sogenanntes Clinical Decision Support System (CDSS) geben, das ein Team von Wissenschaftlern um Dr. Sebastian Försch vom Institut für Pathologie der Universitätsmedizin Mainz aktuell entwickelt. Bei dem CDSS handelt es sich um eine Softwareplattform, die Gewebeabschnitte analysieren soll. Dazu bedient sich die Software sogenannter Deep-Learning-Algorithmen, einer Form von selbstlernenden Programmen.
„Bei den verwendeten Algorithmen handelt es sich um sogenannte tiefe neuronale Netze, die in ihrem Aufbau und in ihrer Funktion biologischen Neuronenverbänden nachempfunden sind“, erklärt Dr. Sebastian Försch. „Damit ein solches Netzwerk tumorverdächtige von gesunden Gewebearealen unterscheiden kann, muss man es an möglichst vielen verschiedenen Beispielen trainieren. Wir sprechen hier von Millionen von Abbildungen, welche wir zusammengetragen haben“, fährt Försch fort. Das CDSS befindet sich demzufolge in einem fortwährenden Lernprozess. Währenddessen überprüft das Netzwerk permanent die Richtigkeit seiner Vorhersage. „Liegt das CDSS falsch, so korrigiert es sich selbst und versucht dann, zu einer besseren Vorhersage zu gelangen. Liegt es hingegen richtig, so steuert es auf die optimale Lösung zu“, so Försch.
Täglich werden am Institut für Pathologie der Universitätsmedizin Mainz bis zu 1000 Gewebeschnitte unter dem Mikroskop begutachtet. Bisher verwenden die Fachärzte für Pathologie hierzu überwiegend analoge Lichtmikroskope. Doch eine zunehmende Digitalisierung des pathologischen Untersuchungsguts macht dieses für eine automatisierte, computerbasierte Analyse zugänglich, welche in den alltäglichen Routineworkflow integriert werden könnte.
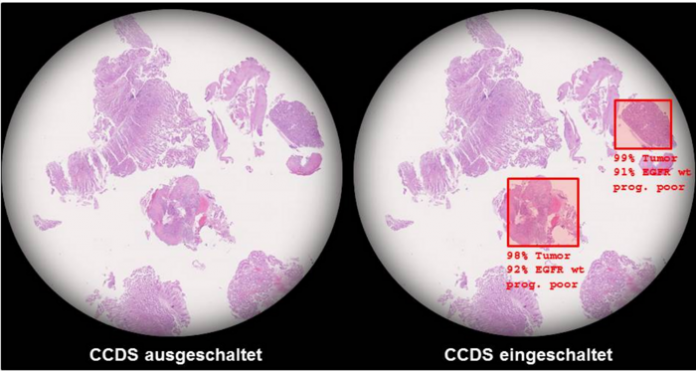
Dabei lässt sich das Ergebnis einer mittels des CDSS durchgeführten Gewebeanalyse potentiell auch visualisieren. Dabei kann man sich zunutze machen, dass das System nicht nur eine Aussage darüber trifft, ob ein Tumor vorliegt oder nicht, sondern auch, wie sicher sich das System bei seiner Aussage ist. „So könnte man diese Wahrscheinlichkeit in Farben umwandeln und das Gewebe ´digital´ einfärben – beispielsweise ein dezentes Grün, wenn alles gesund ist, oder ein alarmierendes Rot, wenn der Algorithmus sich sicher ist, dass es sich um einen Tumor handelt“, unterstreicht Försch.
„Der Nutzen einer solchen Softwareplattform wäre gerade für die Pathologie immens. Einerseits würde dieses CDSS schnellere und präzisere Diagnosen erlauben. Andererseits würde es mit einer erheblichen Kostenersparnis einhergehen, solch ein System an einem pathologischen Institut zu verwenden“, ist der Direktor des Instituts für Pathologie Univ.-Prof. Dr. Wilfried Roth überzeugt. „Was dieses Forschungsprojekt so viel versprechend macht, ist, dass das CDSS aus der Pathologie heraus entwickelt wird. Dadurch befinden wir uns in der einmaligen Situation, bereits vom ersten Projekttag an auf spezielle pathologische Anforderungen eingehen zu können“, sagt Dr. Försch.
Neben dem Institut für Pathologie ist noch die Stabsstelle Technologietransfer der Universitätsmedizin Mainz und das Institut für Informatik der Johannes Gutenberg-Universität Mainz (JGU) an dem Projekt beteiligt.
Bildunterzeile: Mögliche Anwendung während der Routinediagnostik: Links – Gewebeproben einer Magenspiegelung ohne visuelle Unterstützung. Rechts – Mögliche Darstellung auffälliger Areale nach automatisierter Analyse. Neben der sogenannten Gewebedignität sollen noch weitere Informationen über molekulare oder klinische Eigenschaften angezeigt werden. (Theoretisches Modell – Entwicklung noch nicht abgeschlossen – Keine klinische Anwendung)
